Elaboração de mapas de distribuição de espécies em R
Comandos básicos para você preparar seu próprio mapa
Dr. Ricardo Perdiz | 21--22 Out 2021 | Dia 01

Sobre mim

Dr. em Botânica (INPA)
Analista de dados
@ricoperdiz
@ricoperdiz
ricardoperdiz.com
ricoperdiz@gmail.com
Meu nome é Ricardo de Oliveira Perdiz. Sou um biólogo com mestrado e doutorado em botânica, minha linha de pesquisa é com a evolução e sistemática da flora Neotropical, em especial a família do palo santo, mirra, breus, e amesclas, conhecida pelo nome científico Burseraceae. Também me dedico à criação de ferramentas para facilitar a análise de dados taxonômicos e produção de monografias taxonômicas.
Sumário de hoje
Introdução básica ao R
Tutorial para elaborar o mapa
Carregar pacotes
Importar dados
Sumário de hoje
Introdução básica ao R
Tutorial para elaborar o mapa
Carregar pacotes
Importar dados
Checagem e limpeza de dados
Versão simplificada do tutorial Obtendo dados e plotando mapas no R
Carregar pacotes
Importar dados
Checagem e limpeza de dados
Criação de variáveis
Plota o mapa
Apresentarei uma versão simplificada do tutorial, SEM A CHECAGEM e LIMPEZA dos DADOS (RISCAR ESSA PARTE).
Essa parte amanhã será explorada no dia 2 do minicurso.
Desejo agora explanar brevemente sobre o que é o R, e passar conhecimentos bem simplificados que são necessários para manipulação de dados em ambiente R.
O que é o R?
R é uma linguagem e ambiente para análise estatística e produção de gráficos de alta qualidade.

O que é o R?
R pode ser utilizado para manipular e analisar dados, criar gráficos, e simulação computacional.
Por que usar o R???
1. É grátis
2. Gráficos excelentes
3. Diversidade de ferramentas (=pacotes) para fazer análises estatísticas
Por que usar o R???
1. É grátis
2. Gráficos excelentes
3. Diversidade de ferramentas (=pacotes) para fazer análises estatísticas
4. Liberdade para criação de funções
Como obter o R?
Visitar a página do projeto: www.r-project.org.
Verificar este tutorial para instalação: https://github.com/ricoperdiz/minicurso-elaboracao-mapa-R/blob/main/extra-ref/Instrucoes_ROPerdiz_instalando_Baixando_R.pdf.
Como é a linguagem do R
- Escrever em R é como escrever em português, inglês etc;
Como é a linguagem do R
Escrever em R é como escrever em português, inglês etc;
Programar em linguagens como R consiste em definir funções e variáveis;
Como é a linguagem do R
Escrever em R é como escrever em português, inglês etc;
Programar em linguagens como R consiste em definir funções e variáveis;
Variáveis podem ser números ou palavras; guardamos esses valores para uso posterior;
Como é a linguagem do R
Escrever em R é como escrever em português, inglês etc;
Programar em linguagens como R consiste em definir funções e variáveis;
Variáveis podem ser números ou palavras; guardamos esses valores para uso posterior;
Funções executam comandos, por meio de argumentos;
Como é a linguagem do R
Escrever em R é como escrever em português, inglês etc;
Programar em linguagens como R consiste em definir funções e variáveis;
Variáveis podem ser números ou palavras; guardamos esses valores para uso posterior;
Funções executam comandos, por meio de argumentos;
Comandos geram resultados.
Variáveis
Variáveis em R são assinaladas utilizando a notação <-.
meunome <- "Ricardo de Oliveira Perdiz"idade <- 17paste(meunome, "tem", idade, "anos")## [1] "Ricardo de Oliveira Perdiz tem 17 anos"Mais exemplos
> string <- 'Olá, Itacoatiara!'> meu_numero <- 25> string## [1] "Olá, Itacoatiara!"> meu_numero## [1] 25> print(string)## [1] "Olá, Itacoatiara!"> print(meu_numero)## [1] 25Funções e argumentos
- Função -> é um elemento em R que requer uma ação do computador. Cumpre determinada tarefa.
Funções e argumentos
Função -> é um elemento em R que requer uma ação do computador. Cumpre determinada tarefa.
Argumento -> especifica ou modifica como uma função trabalha. É especificado dentro de "parênteses" após o nome da função
> seq(from = 0,to = 10,by =1)## [1] 0 1 2 3 4 5 6 7 8 9 10Como é a linguagem do R
Anatomia de uma linha de comando
> x <- sample(1:10,20,replace=TRUE)> x <- sample(1:10, 20, replace = TRUE)> print(x)## [1] 5 4 5 6 2 3 8 1 8 7 10 7 5 6 6 2 1 10 2 3> mean(x)## [1] 5.05Detalhes nomenclaturais em R
- Separador de decimal é ponto (= "."); vírgula separa argumentos
Detalhes nomenclaturais em R
Separador de decimal é ponto (= "."); vírgula separa argumentos
O símbolo de igual "=" atribui valores aos argumentos
Detalhes nomenclaturais em R
Separador de decimal é ponto (= "."); vírgula separa argumentos
O símbolo de igual "=" atribui valores aos argumentos
Nomes de argumentos podem ser omitidos, desde que os valores estejam seguindo a ordem pré-determinada da função. Ver o '?help'
Detalhes nomenclaturais em R
Separador de decimal é ponto (= "."); vírgula separa argumentos
O símbolo de igual "=" atribui valores aos argumentos
Nomes de argumentos podem ser omitidos, desde que os valores estejam seguindo a ordem pré-determinada da função. Ver o '?help'
Aspas guardam texto; parênteses servem para abrir e fechar funções ou vetores; nomes de objetos não podem ter espaços nem símbolos especiais.
O meu melhor amigo é o ? (ou a ajuda do R!)
Ter dúvidas é comum ao programar.
O meu melhor amigo é o ? (ou a ajuda do R!)
Ter dúvidas é comum ao programar.
O R possui um excelente recurso, o ? (ajuda, ou "help").
O meu melhor amigo é o ? (ou a ajuda do R!)
Ter dúvidas é comum ao programar.
O R possui um excelente recurso, o ? (ajuda, ou "help").
Uma das maneiras é digitar "?" + "nomeDaFunção".
?seqViva o poder da linguagem R
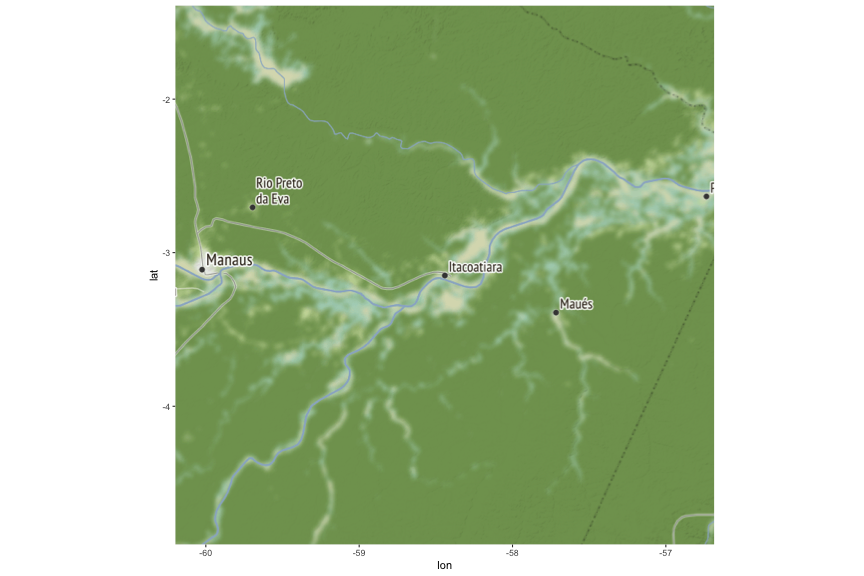
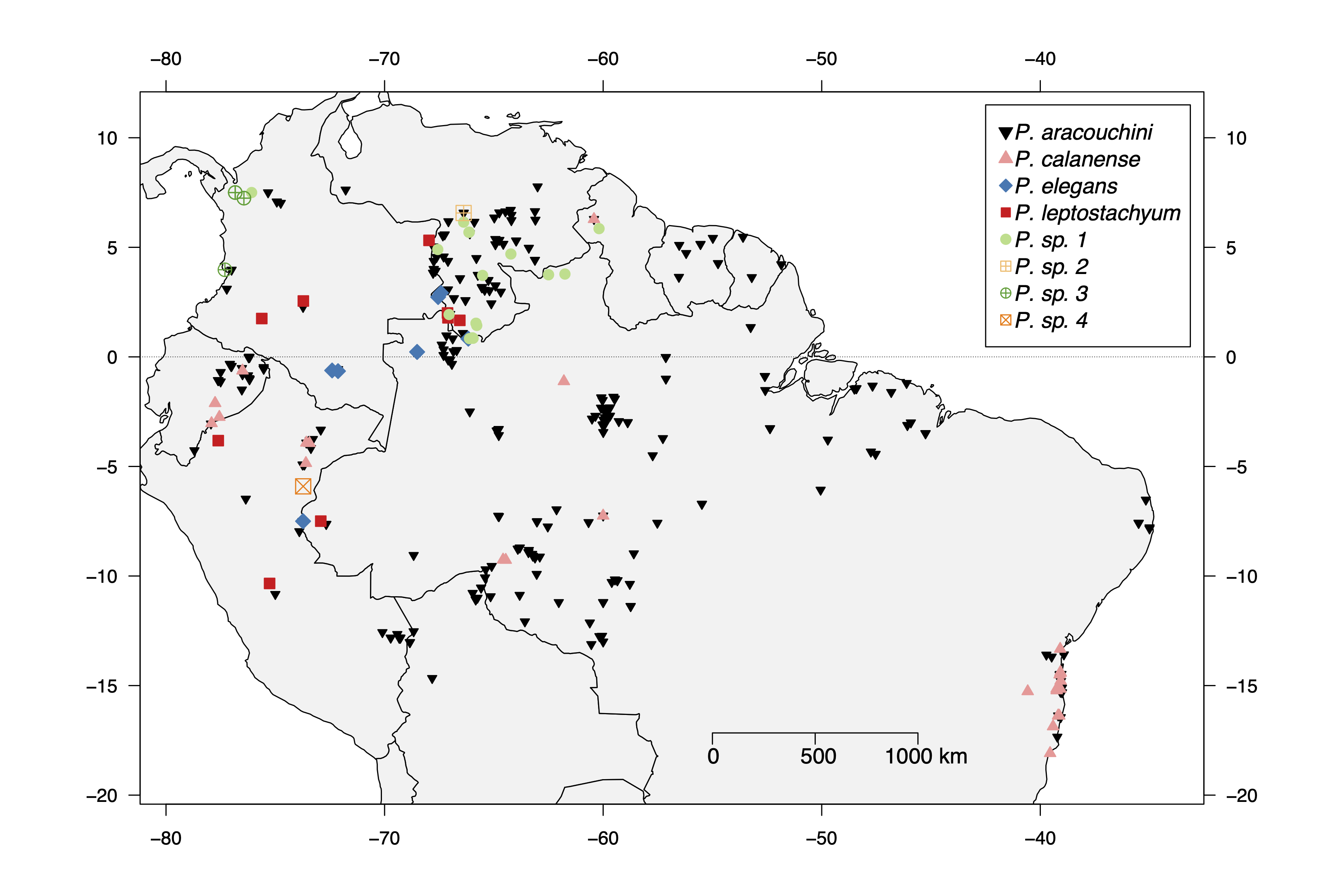
O tutorial é bem simples e visa a produção de um mapa de distribuição geográfica utilizando dados que abrangem a distribuição espacial de duas espécies de Burseraceae, Protium aracouchini (Aubl.) Marchand e P. heptaphyllum (Aubl.) Marchand. A primeira planta faz parte de um complexo de espécies informalmente denominado complexo Protium aracouchini, cuja sistemática e taxonomia vem sendo objeto de estudo em meu doutorado. A segunda espécie, P. heptaphyllum, é objeto de estudo de Gabriel Damasco, colaborador do LABOTAM e aluno de doutorado na Universidade da Califórnia, Berkeley.
Os dados foram baixados do herbário virtual do Jardim Botânico de Nova Iorque que é onde trabalha o especialista em Burseraceae, Dr. Douglas Daly, e cuja coleção de espécimes e dados vêm sendo bem cuidada há algumas décadas. Para esta postagem, filtramos apenas os dados para as duas espécies citadas acima.
Assuntos abordados
(Instalar e) Carregar pacotes
Importação de dados
Checagem de dados
Criação de variáveis
Plotagem do mapa
A postagem original faz uso dos pacotes maps (para plotar o mapa), RColorBrewer (paleta de cores), magrittr (pipe, ou %>%) e dplyr (manipulação de 'dataframes').
Neste minicurso, utilizaremos apenas o pacote maps, a fim de simplificar o aprendizado.
Importando os dados
Os dados estão reunidos em um arquivo chamado dados.csv, disponível em: https://github.com/ricoperdiz/Tutorials/blob/master/R_make_a_map/dados.csv.
Importando os dados
Os dados estão reunidos em um arquivo chamado dados.csv, disponível em: https://github.com/ricoperdiz/Tutorials/blob/master/R_make_a_map/dados.csv.
As informações estão separadas por tabulação e codificadas em UTF-8.
Importando os dados
Os dados estão reunidos em um arquivo chamado dados.csv, disponível em: https://github.com/ricoperdiz/Tutorials/blob/master/R_make_a_map/dados.csv.
As informações estão separadas por tabulação e codificadas em UTF-8.
Você pode baixar os dados direto da página do GitHub ou fazer uso do comando abaixo para ler diretamente os dados da rede.
protium <- read.table( file = url( "https://raw.githubusercontent.com/ricoperdiz/Tutorials/master/R_make_a_map/dados.csv" ), header = TRUE, as.is = TRUE, sep = '\t', dec = '.' )Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Argumentos
file - Local do arquivo
Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Argumentos
file - Local do arquivo
header - Tem cabeçalho ou não?
Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Argumentos
file - Local do arquivo
header - Tem cabeçalho ou não?
as.is - Classes dos vetores. Deixa como está ou faz alguma conversão?
Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Argumentos
file - Local do arquivo
header - Tem cabeçalho ou não?
as.is - Classes dos vetores. Deixa como está ou faz alguma conversão?
sep - Separador de texto.
Destrinchando o read.table
read.table() - Função básica para ler arquivos no R
Argumentos
file - Local do arquivo
header - Tem cabeçalho ou não?
as.is - Classes dos vetores. Deixa como está ou faz alguma conversão?
sep - Separador de texto.
dec - Separador de decimal.
Checagem inicial dos dados
| recordedBy | recordNumber | decimalLatitude | decimalLongitude | identifiedBy | specificEpithet | ID | Species |
|---|
recordedBy | recordNumber | decimalLatitude | decimalLongitude | identifiedBy | specificEpithet | ID | Species | |
|---|---|---|---|---|---|---|---|---|
| 1 | A_A_de_Oliveira | 278 | -2.31537 | -59.6855 | D. C. Daly | aracouchini | A_A_de_Oliveira_278 | Protium aracouchini |
| 2 | A_B_Anderson | 214 | -3.18524 | -60.1302 | D. C. Daly | heptaphyllum | A_B_Anderson_214 | Protium heptaphyllum |
| 3 | A_C_Londoño | 1486 | -0.57 | -72.13 | D. C. Daly | aracouchini | A_C_Londoño_1486 | Protium aracouchini |
| 4 | A_C_Londoño | 493 | -0.57 | -72.13 | D. C. Daly | aracouchini | A_C_Londoño_493 | Protium aracouchini |
| 5 | A_C_Weber | s_n | 0.3 | -66.7 | D. C. Daly | aracouchini | A_C_Weber_s_n | Protium aracouchini |
| 6 | A_Castillo | 1609 | 6.42 | -67.42 | D. C. Daly | heptaphyllum | A_Castillo_1609 | Protium heptaphyllum |
| 7 | A_Castillo | 2297 | 6.42 | -67.42 | D. C. Daly | heptaphyllum | A_Castillo_2297 | Protium heptaphyllum |
| 8 | A_Castillo | 2372 | 6.42 | -67.42 | D. C. Daly | heptaphyllum | A_Castillo_2372 | Protium heptaphyllum |
| 9 | A_Castillo | 4611 | 4.975 | -67.667 | D. C. Daly | heptaphyllum | A_Castillo_4611 | Protium heptaphyllum |
| 10 | A_Castillo | 4650 | 4.975 | -67.667 | D. C. Daly | heptaphyllum | A_Castillo_4650 | Protium heptaphyllum |
Checagem e limpeza dos dados
Existência de valores vazios; em caso positivo, devemos eliminá-los;
Checagem e limpeza dos dados
Existência de valores vazios; em caso positivo, devemos eliminá-los;
Confiabilidade dos valores de latitude e longitude
Checagem e limpeza dos dados
Existência de valores vazios; em caso positivo, devemos eliminá-los;
Confiabilidade dos valores de latitude e longitude
Valores duplicados?
Há valores vazios?
Dados de Latitude e latitude, às vezes, por diversos fatores, há troca de sinais (negativos e positivos) ocasionando equívocos quanto à ocorrência exata da amostra. Se for percebido algo assim, é bom checar os dados e buscar corrigí-los.
Há valores duplicados?
Um pouco de classe de objetos
Nossa tabela, após a importação, é chamada de data.frame.
class(protium)## [1] "data.frame"dim(protium)## [1] 667 8Acessando vetores em (ou partes de) um data.frame
Utilizamos o "$"
head(protium$recordedBy)## [1] "A_A_de_Oliveira" "A_B_Anderson" "A_C_Londoño" ## [4] "A_C_Londoño" "A_C_Weber" "A_Castillo"Acessando vetores em (ou partes de) um data.frame
Ou então colchetes, com o nome da coluna dentro de aspas, dentro dos colchetes
head(protium[, "recordedBy"])## [1] "A_A_de_Oliveira" "A_B_Anderson" "A_C_Londoño" ## [4] "A_C_Londoño" "A_C_Weber" "A_Castillo"Cria variáveis para plotar o mapa
Nomes de espécies
unique(protium$Species)## [1] "Protium aracouchini" "Protium heptaphyllum"spp <- unique(protium$Species)spp## [1] "Protium aracouchini" "Protium heptaphyllum"Variáveis - Vetor de cores para utilizar no mapa
Para cada espécie, uma cor!
Vamos conhecer o famoso "if else", ou em português:
Se ..., caso contrário ...
Variáveis - Vetor de cores para utilizar no mapa
Para cada espécie, uma cor!
Vamos conhecer o famoso "if else", ou em português:
Se ..., caso contrário ...
head(ifelse(protium$Species == spp[1], 'red','black'), 50)## [1] "red" "black" "red" "red" "red" "black" "black" "black"## [9] "black" "black" "black" "black" "black" "red" "red" "red" ## [17] "red" "black" "black" "red" "red" "red" "black" "red" ## [25] "black" "red" "black" "black" "red" "red" "red" "black"## [33] "red" "red" "black" "red" "red" "red" "red" "black"## [41] "black" "black" "red" "black" "black" "red" "black" "black"## [49] "red" "black"Destrinchando o ifelse
spp[1]## [1] "Protium aracouchini"head(protium$Species == spp[1], 10)## [1] TRUE FALSE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSEVariáveis - Vetor de cores para utilizar no mapa
Para cada espécie, uma cor!
cores.map <- ifelse(protium$Species == spp[1], 'red','black')head(cores.map, 50)## [1] "red" "black" "red" "red" "red" "black" "black" "black"## [9] "black" "black" "black" "black" "black" "red" "red" "red" ## [17] "red" "black" "black" "red" "red" "red" "black" "red" ## [25] "black" "red" "black" "black" "red" "red" "red" "black"## [33] "red" "red" "black" "red" "red" "red" "red" "black"## [41] "black" "black" "red" "black" "black" "red" "black" "black"## [49] "red" "black"Fim do dia 1!
Amanhã, dia 22 de outubro, continuaremos!

Dúvidas? Entrem em contato!
@ricoperdiz
@ricoperdiz
ricardoperdiz.com
ricoperdiz@gmail.com